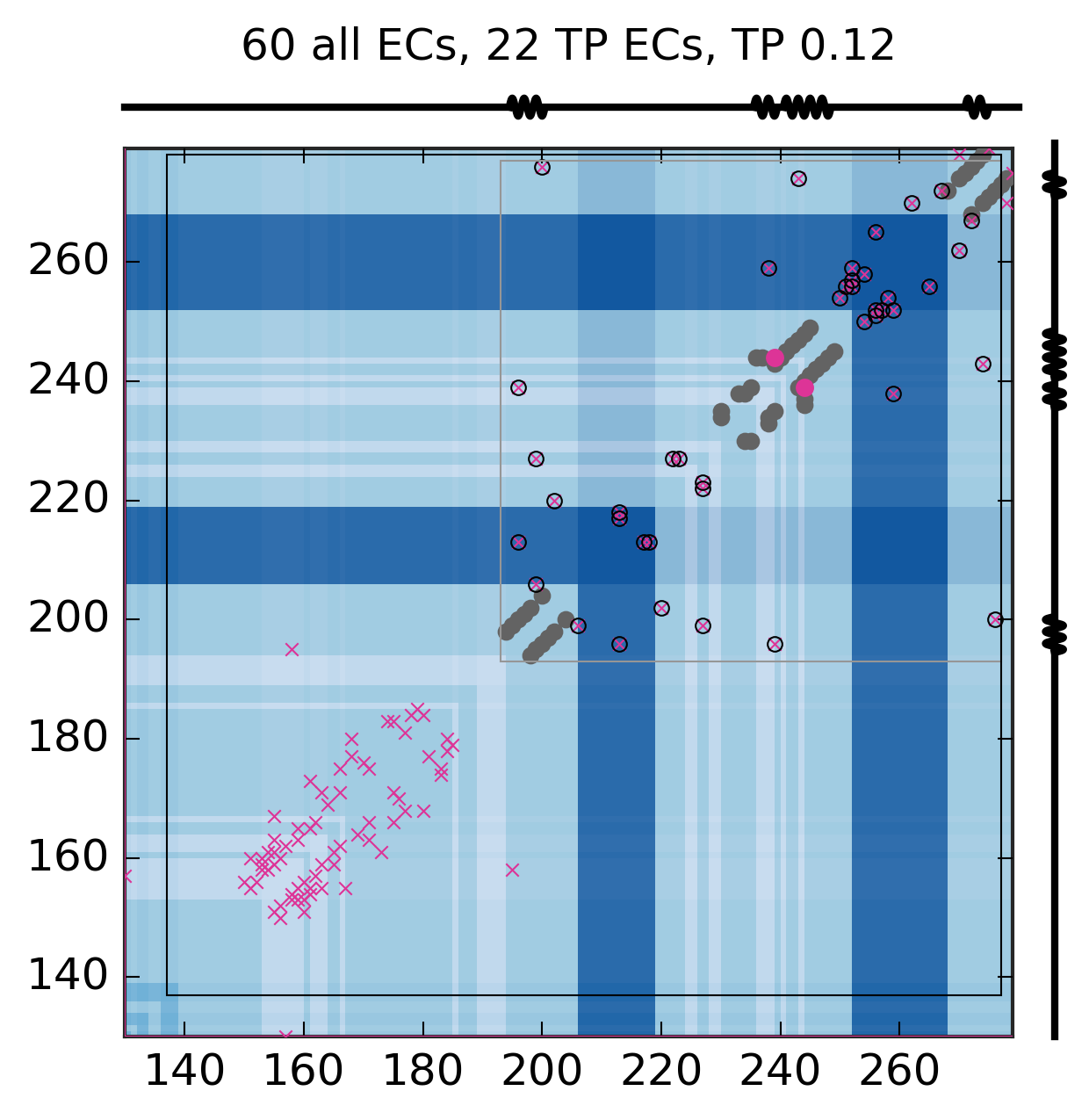
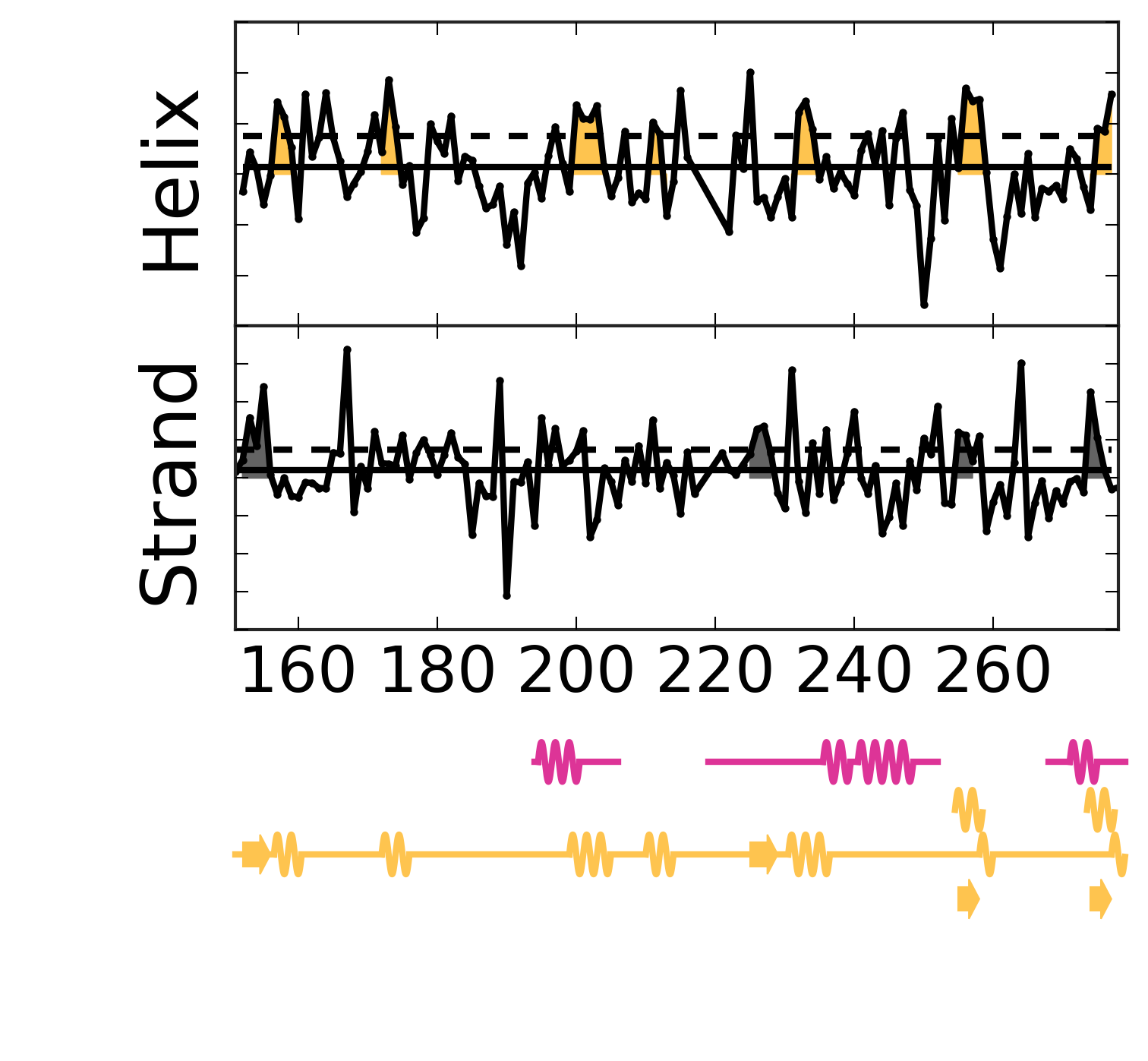
Evolutionary Coupling Analysis
Predicted and experimental contacts
Secondary structure from ECs
EC score distribution and threshold
Top ECs
| Rank |
Residue 1 |
Amino acid 1 |
Residue 2 |
Amino acid 2 |
EC score |
| 1 |
196 |
D |
239 |
I |
0.24 |
| 2 |
159 |
S |
163 |
V |
0.22 |
| 3 |
155 |
V |
159 |
S |
0.22 |
| 4 |
166 |
K |
171 |
S |
0.21 |
| 5 |
213 |
P |
217 |
G |
0.20 |
| 6 |
174 |
P |
183 |
N |
0.20 |
| 7 |
162 |
V |
166 |
K |
0.20 |
| 8 |
179 |
E |
185 |
L |
0.19 |
| 9 |
154 |
T |
161 |
K |
0.19 |
| 10 |
153 |
V |
158 |
A |
0.19 |
| 11 |
254 |
T |
258 |
P |
0.18 |
| 12 |
223 |
P |
227 |
T |
0.18 |
| 13 |
250 |
N |
254 |
T |
0.18 |
| 14 |
151 |
G |
155 |
V |
0.17 |
| 15 |
152 |
G |
156 |
A |
0.17 |
| 16 |
270 |
I |
278 |
T |
0.17 |
| 17 |
252 |
A |
257 |
L |
0.17 |
| 18 |
155 |
V |
167 |
K |
0.17 |
| 19 |
180 |
S |
184 |
K |
0.16 |
| 20 |
155 |
V |
161 |
K |
0.16 |
| 21 |
252 |
A |
256 |
P |
0.16 |
| 22 |
158 |
A |
195 |
L |
0.16 |
| 23 |
251 |
E |
256 |
P |
0.16 |
| 24 |
239 |
I |
244 |
R |
0.16 |
| 25 |
267 |
D |
272 |
A |
0.16 |
| 26 |
153 |
V |
160 |
D |
0.15 |
| 27 |
178 |
M |
184 |
K |
0.15 |
| 28 |
213 |
P |
218 |
P |
0.15 |
| 29 |
177 |
P |
181 |
T |
0.15 |
| 30 |
156 |
A |
160 |
D |
0.15 |
| 31 |
199 |
I |
206 |
E |
0.15 |
| 32 |
199 |
I |
227 |
T |
0.15 |
| 33 |
171 |
S |
175 |
T |
0.15 |
| 34 |
262 |
K |
270 |
I |
0.15 |
| 35 |
150 |
A |
156 |
A |
0.15 |
| 36 |
275 |
S |
279 |
C |
0.15 |
| 37 |
130 |
K |
157 |
T |
0.15 |
| 38 |
161 |
K |
165 |
K |
0.15 |
| 39 |
222 |
D |
227 |
T |
0.15 |
| 40 |
238 |
T |
259 |
D |
0.15 |
| 41 |
200 |
D |
276 |
D |
0.15 |
| 42 |
155 |
V |
163 |
V |
0.15 |
| 43 |
196 |
D |
213 |
P |
0.14 |
| 44 |
157 |
T |
162 |
V |
0.14 |
| 45 |
153 |
V |
159 |
S |
0.14 |
| 46 |
252 |
A |
259 |
D |
0.14 |
| 47 |
202 |
L |
220 |
V |
0.14 |
| 48 |
243 |
Y |
274 |
S |
0.14 |
| 49 |
163 |
V |
171 |
S |
0.14 |
| 50 |
166 |
K |
175 |
T |
0.14 |
| 51 |
161 |
K |
173 |
T |
0.14 |
| 52 |
175 |
T |
183 |
N |
0.14 |
| 53 |
170 |
K |
176 |
L |
0.14 |
| 54 |
168 |
E |
180 |
S |
0.14 |
| 55 |
256 |
P |
265 |
G |
0.14 |
| 56 |
151 |
G |
160 |
D |
0.14 |
| 57 |
164 |
V |
169 |
K |
0.14 |
| 58 |
154 |
T |
158 |
A |
0.14 |
| 59 |
168 |
E |
177 |
P |
0.14 |
| 60 |
159 |
S |
165 |
K |
0.14 |
Alignment robustness analysis
First most common residue correlation
Second most common residue correlation